应用行业:医学&药学 技术:石英晶体微天平 材料:冠状病毒,COVID-19
新型冠状病毒(COVID-19)已经成为一个全球性的大流行,从政府基础设施和市场,到我们日常生活中彼此之间的互动,COVID-19对我们生活的各个层面都造成了极大的破坏。与之前的寨卡病毒、急性呼吸综合征(SARS)以及H1N1(禽流感)类似,COVID-19需要一个专门的多管齐下的方法来遏制其爆发并最终击败它。通过保持社交距离和洗手等我们每个人都能做到的方法可以遏制和阻挡病毒传播[1],但是这些方法需要严格和谨慎的执行才能有效。长期的解决方案比如疫苗开发需要巨大的投资及研究来了解病毒如何攻击身体和其细胞的机制。
病毒通过侵入并劫持细胞合成新的病毒进行复制。疫苗通过率先将身体的免疫系统暴露于灭活/减活形式的病毒或者病毒的关键组成部分如其被膜或者表面抗原来阻止病毒的复制。暴露之后,身体在此后再次遭遇此病毒时能够识别并中和病毒颗粒。要确定灭活或分离哪一部分的病毒以形成疫苗,需要对它的结构进行仔细研究,并发现如何以灵敏和特异的方式与病毒结合。
开发准确的检测方法来确定一个人是否已经被感染是保障人体健康的一个补充性步骤。病毒检测方法包括抗体检测、DNA/RNA检测、抗原检测。这些方法的成功实施需要了解究竟哪种病毒组分能够被最特异和灵敏的识别出来。对于有些病毒,涉及寻找感染病例产生的抗体或者病毒抗原。对于另外一些病毒,则将需要提取病毒的DNA或者RNA来匹配其互补链[2]。任何一种策略都需要一个高内涵、高灵敏的技术来准确、可重复性地确定这些分子的结合。
QSense耗散型石英晶体微天平技术是一种实时在线、无需标记的测量生物分子吸附的技术,已经有几十年的使用历史。此技术利用一个石英芯片,当加载电压时能够在芯片共振频率处产生振荡。当有质量负载在芯片上时,其振动频率将会降低,频率的降低幅度直接与负载的重量多少相关。除了频率变化,QSense技术还能测量耗散,即当施加电压停止后芯片振荡静止下来的快慢。振荡静止的快慢与负载物质的刚性程度相关。由于更加柔性的材料倾向于更容易消耗掉动能,所以与柔性物质相比,刚性的物质负载趋向于更加缓慢的振荡停止。QSense技术具有ng/cm2量级的灵敏度,适合测量病毒、DNA和蛋白质-蛋白质相互作用。如需了解更多关于QSENSE技术的细节,请浏览我们的技术页面。
支撑脂双层膜(SLB)作为模型体系研究病毒相互作用
长期以来,以QCM-D平台作为一种芯片技术来研究生物分子相互作用的适用性被广泛接受。将一个受体分子吸附或者固定于表面后,其与目标配体的相互作用就能够被监测到。因为支撑脂双层膜可以模拟细胞膜,所以它在QCM-D研究中被用作一种常规基底。这些脂质双层膜通常以脂囊泡的形式先合成出来。QSense技术的质量测量属性使其成为追踪脂质从囊泡结构到形成脂质双层结构的理想手段。根据表面化学,这些囊泡有可能以完整形式吸附于芯片表面或者破裂进而形成预期的脂双层[3]。QCM-D可以清晰地追踪从起始吸附开始后的频率下降,及随后因为囊泡内部耦合溶剂的损失导致的频率上升。因为起始吸附是一个柔软的高含水的质量,生意耗散一开始上升,随后因为囊泡铺展形成刚性的双层而耗散下降。
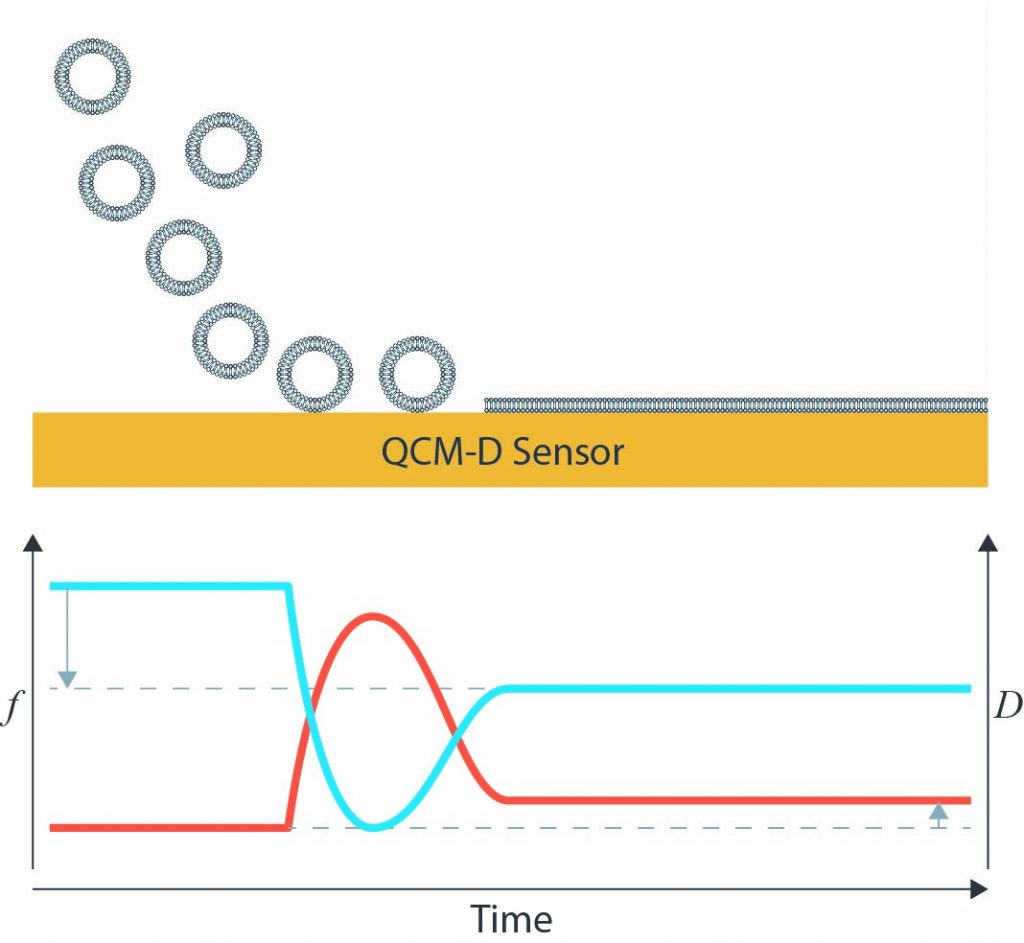
图1 – 囊泡吸附于QCM-D表面形成支撑脂双层和其典型的频率(蓝色)和耗散(橘色)响应。
研究者可以将与病毒蛋白或者外壳相互作用的蛋白质插入其中来利用这些双层。Cho等研究了来自于丙型肝炎病毒的NS5A蛋白质结合于Huh7 (肝细胞)生成膜[4]。他们假设NS5A蛋白中的两亲螺旋(AH)结合域是肝细胞与丙型肝炎蛋白之间的结合。他们用QCM-D观察到NS5A蛋白吸附到Huh7膜上,当AH结构域被其他结构取代时,这种结合不再发生。此外,当Huh7膜暴露于表面不含蛋白的胰蛋白酶时,NS5A蛋白也不再结合。这表明AH结合位点与肝细胞表面某些表面蛋白存在特异性相互作用。几年后,该课题组将对NS5A与肝细胞研究进一步缩小到磷脂酰肌醇4,5-二磷酸(PI(4,5)P2与NS5A蛋白中的BAAP域之间的相互作用。通过比较PI(4,5)P2的变异体和抑制剂[5]的 QCM-D响应,这些结果再次得到证实。
通过表面化学手段固定蛋白质
因为其重量分析性质QSense技术还有另外一个主要优势,任何可以被涂层于石英芯片上的表面都可以用来作为一种基质。这使其拥有宽广的应用空间,可覆盖的应用如电化学、膜污染、药物-包装相互作用等等。对于病毒研究,表面化学技术可以用来制作如图2那样的生物芯片型表面。一个硫醇化的聚乙二醇(PEG)表面可以用来吸附一层生物素-亲和链霉亲和素(biotin-streptavidin)层,然后以层层组装(layer-by-layer)的方式构建其他感兴趣的均匀分布的抗体。随后这个抗体可以作为特定分子的检测器,此案例中,特定分子是牛血清蛋白(BSA). 整个相互作用过程可以通过QSense技术如预期那样进行原位监测。另外,此表面可以如图3所示那样进行再生和重建。如图3左所示,此芯片再生过程执行了五个步骤,每一步都重复性地将频率和耗散值恢复至一个稳定的基线。图3右中,对每一个试验的每一步和再生循环进行了更加详细的展示,这里可以清晰地看到BSA与BSA抗体之间的相互作用。随后通入NaOH流过芯片表面,清除抗-BSA和BSA. 在每一步试验之间都有一个使用缓冲液的润洗步骤来移除松散结合的分子。
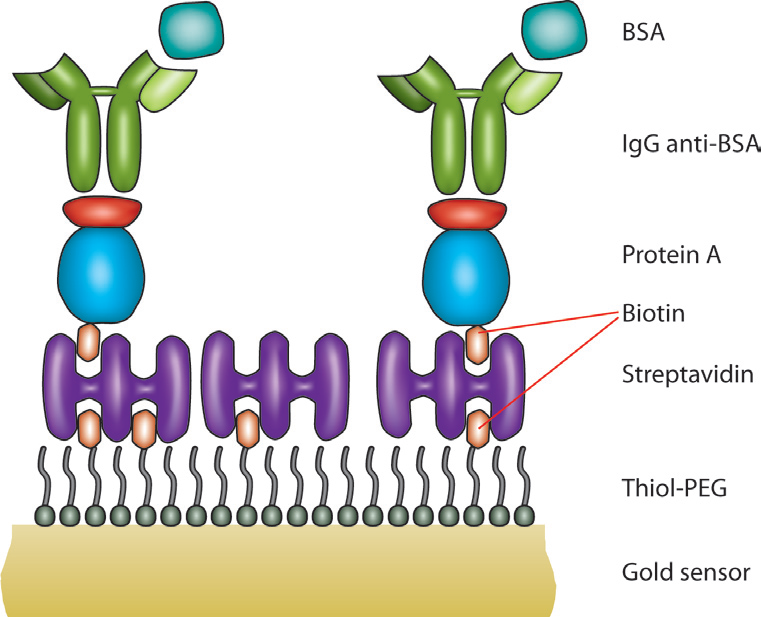
图2-通过生物素-链霉亲和素层构建的BSA捕获表面示意图
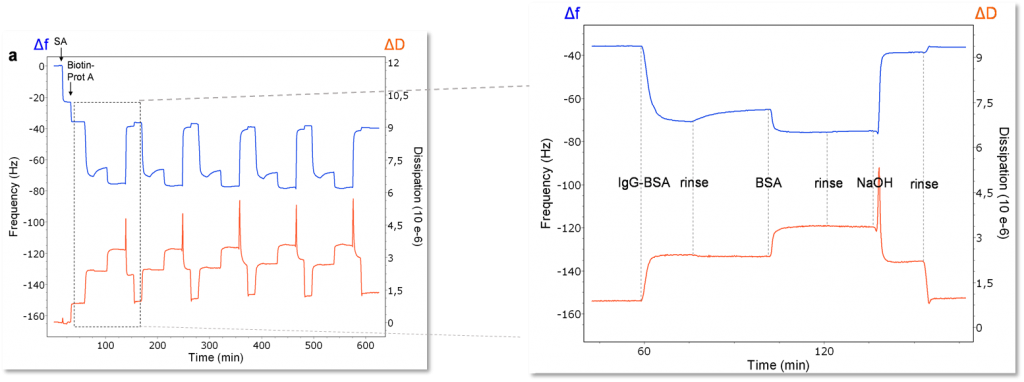
图3 – 捕获表面生成的原始F和D数据(L)和结合/再生步骤的放大图
通过表面化学的方法可以将分子以共价键方式固定于表面。Lee等人描述了一种将NTA(3,3′dithiobis[N-(5-amino-5-(carboxypentyl) propionamide)-N,N′-diacetic acid] dihyrochloride)固定于表面的方法。随后NTA与HIV-1的包膜蛋白gp120通过六聚组氨酸标记(his-tag)相互作用而固定此蛋白[6]。图4为此实验设计的示意图。他们随后将gp120暴露于四种不同的配体分子- sCD4, HNG-156, BMS-806, 和 NBD-556。除了BMS-806之外,每个配体分子的耦合都观察到了由于gp120构象变化致使的分子链收缩。实验过程中观测到耗散上升和频率下降,表明质量有损失。作者提到QCM-D响应与蛋白质同步的分子链收缩和耦合水的释放相一致。另一方面,BMS-606分子显示了其与gp120暂时的结合并且随后被冲洗掉。
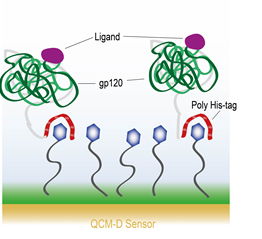
图4 – NTA功能化的金表面与HIV-1 gp120蛋白通过组氨酸标记(his-tag)相结合示意图。
目标配体随后与gp120相互作用。
DNA杂交
一些QCM-D研究检测了基于石英传感器的DNA杂交。 Gunnarsson 等人研究了通过生物素化 PLL-g-PEG 分子固定 15单体单元的DNA分子,随后此DNA分子与连接到脂质囊泡的 30单体单元互补链DNA的结合特异性问题 [7]。然后他们在30单体单元DNA链上制造了一个非匹配碱基,观察发现结合到表面的囊泡减少了大约 60%。
Rawle等人使用了一种稍微不同的方法检查DNA杂交。他们没有使用生物素化的捕获表面来固定 DNA,而是使用了一个带正电荷的聚乙烯亚胺(PEI)电解质层作为带电表面,让 DNA 通过静电结合到表面 [8]。在吸附变性 DNA 溶液后,他们观察到初始频率急剧变化,然后出现第二步较慢的频率变化,这表明发生了两步吸附。他们将这种较慢吸附的转变归因于吸附在传感器表面的 DNA 链的杂交。
结语
由于其对小分子的高灵敏度、灵活的传感器构建以及实时在线、无须标记的传感特质,QCM-D是希望扩展其研究能力以仔细检测病毒和病毒颗粒与其目标分子之间相互作用机制的研究人员的绝佳选择。利用这项技术,能够确定特异性结合信息。另外,还可以辨别关于蛋白质或分子构象的生物物理细节。
参考文献
[1] Center for Disease Control and Prevention, https://www.cdc.gov/coronavirus/2019-ncov/prepare/prevention.html, 2020.
[2] World Health Organization, “Vaccine Safety Basics,” https://vaccine-safety-training.org/home.html, 2020.
[3] C. . Keller and B. H. Kasemo, “Surface Specific Kinetics of Lipid Vesicle Adsorption Measured with a Quartz Crystal Microbalance,” Biophysical Journal, vol. 75, no. 3, pp. 1397-1402, 1998.
[4] N.-J. . Cho, K. H. Cheong, C. . Lee, C. W. Frank and J. S. Glenn, “Binding Dynamics of Hepatitis C Virus’ NS5A Amphipathic Peptide to Cell and Model Membranes,” Journal of Virology, vol. 81, no. 12, pp. 6682-6689, 2007.
[5] N.-J. . Cho, C. . Lee, P. S. Pang, E. A. Pham, B. . Fram, K. . Nguyen, A. . Xiong, E. H. Sklan, M. . Elazar, E. S. Koytak, C. . Kersten, K. K. Kanazawa, C. W. Frank, J. S. Glenn and J. S. Glenn, “Phosphatidylinositol 4,5-Bisphosphate Is an HCV NS5A Ligand and Mediates Replication of the Viral Genome,” Gastroenterology, vol. 148, no. 3, pp. 616-625, 2015.
[6] H.-S. . Lee, M. . Contarino, M. . Umashankara, A. . Schön, E. I. Freire, A. B. Smith, I. M. Chaiken and L. S. Penn, “Use of the quartz crystal microbalance to monitor ligand-induced conformational rearrangements in HIV-1 envelope protein gp120,” Analytical and Bioanalytical Chemistry, vol. 396, no. 3, pp. 1143-1152, 2010.
[7] A. Gunnarsson, P. Jonsson, R. Marie, J. O. Tegenfeldt and F. Hook, “Single-Molecule Detection and Mismatch Discrimination of Unlabeled DNA Targets,” Nano Letters, vol. 8, no. 1, pp. 183-188, 2008.
[8] R. J. Rawle, C. D. Selassie and M. S. Johal, “Creation of mammalian single- and double-stranded DNA surfaces: a real-time QCM-D study.,” Langmuir, vol. 23, no. 19, pp. 9563-9566, 2007.
如果您需要更多应用信息或者推文的PDF文档,请点击这里给我们留言。